
本实验中,研究人员采用了一种合理的方法来设计阳离子脂质,并制备用于递送小干扰RNA(siRNA)的配方。以稳定核酸脂质颗粒(SNALP)的关键脂质成分1,2-二甲醇氧基-3-二甲基氨基bing烷(DLinDMA)为例,研究人员利用所提出的可电离阳离子脂质的体内作用机制来指导设计具有优异递送能力的基于DLinDMA结构拓展的脂质。通过筛选后,对性能zui好的脂质(DLin-KC2-DMA),进行了SNALP配制和表征,并在啮齿类动物和非人灵长类动物中分别在低至0.01 mg/kg和0.1 mg/kg的siRNA剂量下,证明具有体内活性。研究发现,DLin-KC2-DMA介导的基因沉默比以前关于体内内源性肝脏基因沉默的报道有了实质性的改进。
表1.1 基于DLin-DMA的结构设计
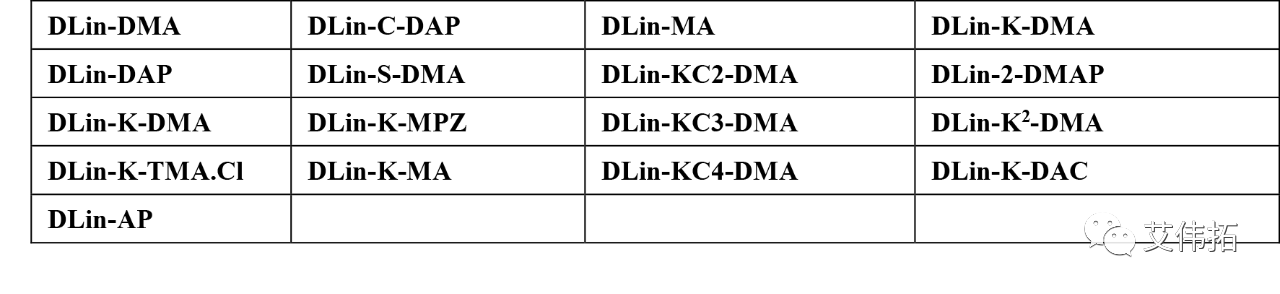
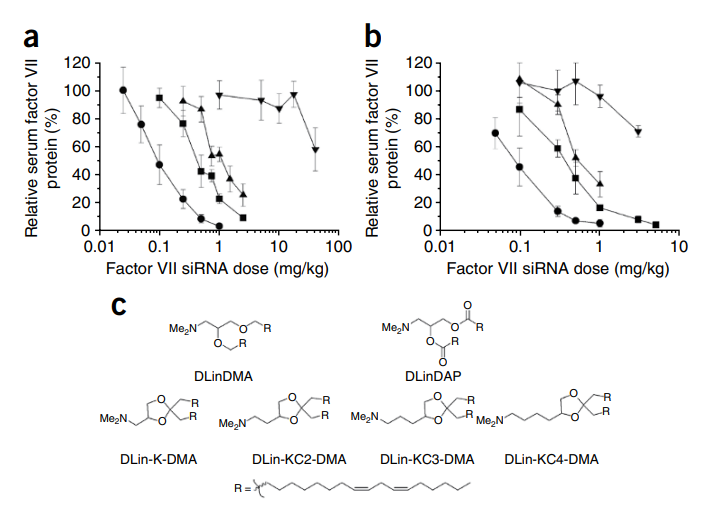
图1.1. 新型阳离子脂质的体内评价
(a). DLinDAP的沉默活性(▼), DLinDMA(▲), DLin-K-DMA(■) 和DLin KC2-DMA(●)在小鼠FVII模型中的筛选。所有LNP siRNA系统都是使用预成型囊泡(PFV)方法制备的,由可电离的阳离子脂质、DSPC、胆固醇和PEG脂质(40:10:40:10mol/mol)组成,FVII siRNA/总脂质的比例约为0.05(wt/wt)。
(b). 头群扩展对DLin-K-DMA活性的影响。DLin-K-DMA(■)在DMA头基和缩酮环连接体之间添加了额外的亚甲基,以生成DLin-KC2-DMA(●),DLin-KC3-DMA(▲)和DLin-KC4-DMA(▼)在小鼠因子VII模型中评估每种脂质的制剂活性。
表1.2 含有新型脂质的LNPs的生物物理参数和体内活性

鉴于正电荷在指导脂质设计的作用机制假说中的重要性,在DLin-K-DMA作为新的基准脂质的综述中研究了胺基头基结构变化的影响。进行了一系列头基修饰、表征和测试,以探索大小、酸离解常数和可电离基团数量的影响(补充合成2和补充表2)。所测试的pai嗪基、ma啉基、三甲基氨基或双二甲基氨基修饰并不优于DLin-K-DMA的基准二甲基氨基头基。作为附加参数,通过引入额外的亚甲基来改变二甲基氨基和二氧wu环连接体之间的距离。该距离可以影响胺头基的pKa以及电荷呈现相对于脂质双层界面的距离和柔性。相对于DLin-K-DMA,将单个额外的亚甲基插入头部基团(DLin-KC2-DMA)产生效力的显著增加。在FVII模型中,这种脂质的ED50约为0.1 mg/kg,使其效力比DLin-K-DMA高4倍,比DLinDMA基准高10倍。然而,具有额外亚甲基的系链的进一步延伸显著降低了活性,DLin-KC3-DMA的ED50约为0.6 mg/kg,DLin-KC4-DMA的ED50>3 mg/kg。
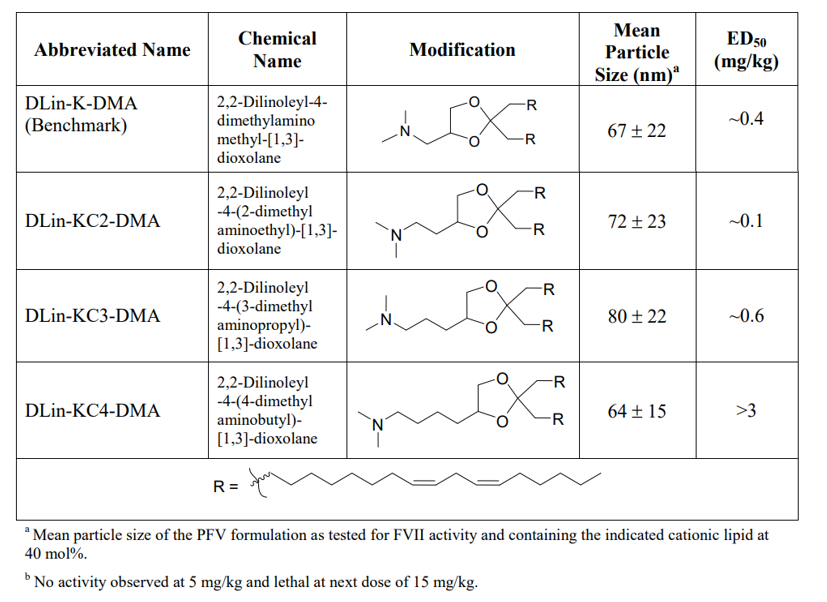
图1.2. 基于DLin-K-DMA,延长头部基团碳链长度对粒径及ED50的影响
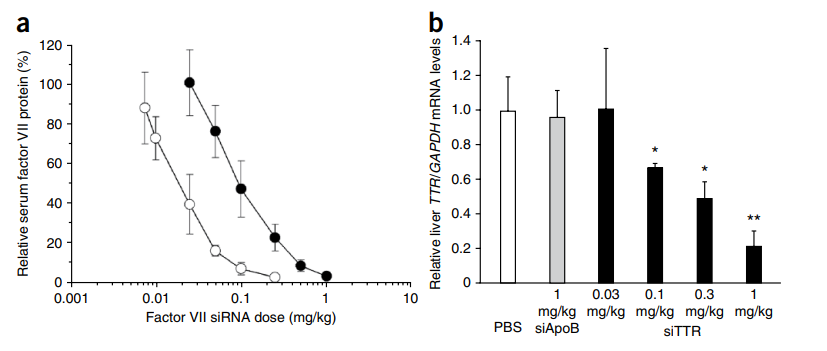
图1.3. KC2-SNALP对啮齿类动物和非人类灵长类动物的疗效
(a) 相对于在小鼠中测试的初始筛选制剂,KC2-SNALP的疗效得到改善。KC2-SNALP的体内疗效(○) 与小鼠因子VII模型中未优化的DLin-KC2-DMA筛选(即PFV)制剂(●)的结果进行比较。数据点以PBS对照动物的百分比表示,代表组平均值(n=5)±s.d.(b)KC2-SNALP在非人灵长类动物中的疗效。食蟹猴(每组n=3)接受0.03、0.1、0.3或1 mg/kg siTTR,或1 mg/kg在KC2-SNALP或PBS中配制的siApoB的总剂量,通过头静脉静脉输注15分钟(5 ml/kg)。给药后48小时对动物实施an乐死。在肝脏样品中测定TTR mRNA水平相对于GAPDH mRNA水平。数据点代表组平均值±s.d.*,P<0.05;**,P<0.005。
表1.3 含有KC2的SNALP的体内活性
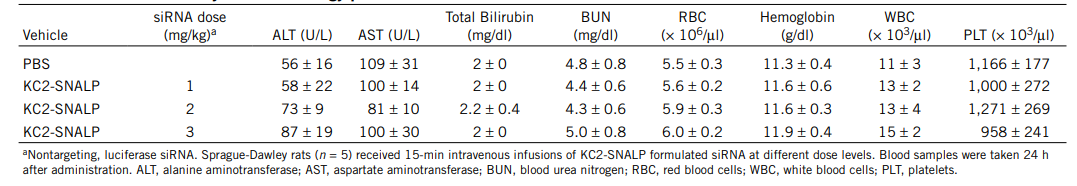
总结,研究人员将一种合理的方法应用于新型阳离子脂质的设计,这些脂质被筛选用于基于LNP的siRNA递送系统。脂质结构分为三个主要功能元件:烷基链、连接基和头基。以DLinDMA为起点,通过保持其他两个元素不变,以系统的方式研究了这些元素中每一个的影响。首先,建立了烷基链,然后改变了连接体,最后,探索了不同的头基结构。使用这种方法,描述了可离子化阳离子脂质的重要结构-活性考虑因素,并鉴定了相对于DLinDMA基准具有改进活性的脂质。性能zui好的脂质(DLin-KC2-DMA)的SNALP制剂在啮齿类和非人类灵长类动物中都具有良好的耐受性,并且在啮齿类动物中在低至0.01mg/kg的siRNA剂量下表现出体内活性,并且在非人类灵长目动物中表现出治疗显著基因(TTR)的沉默。尽管目前的工作范围仅限于体内肝脏递送,但与之前关于LNP siRNA介导的非人灵长类动物沉默的报道相比,这项工作中实现的TTR沉默(ED50~0.3 mg/kg)代表了活性的显著提高
试验2
MC3最大限度地提高siRNA脂质纳米颗粒在体内用于肝脏基因沉默的效力[2]
pKa的考察
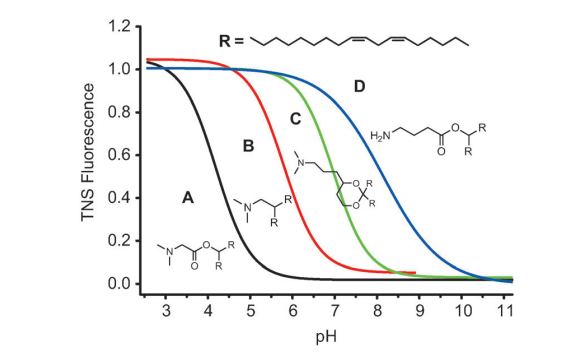
图2.1.TNS荧光法测定pKa
如图2所示,四种可电离阳离子脂质的pKa的测定实例。pKa的取值,定义为在一半最大荧光强度下的pH值。(A). 14,pKa=4.17;(B). 52,pKa=5.73;(C). 19,pKa=6.95;(D). 48,pKa=8.12。
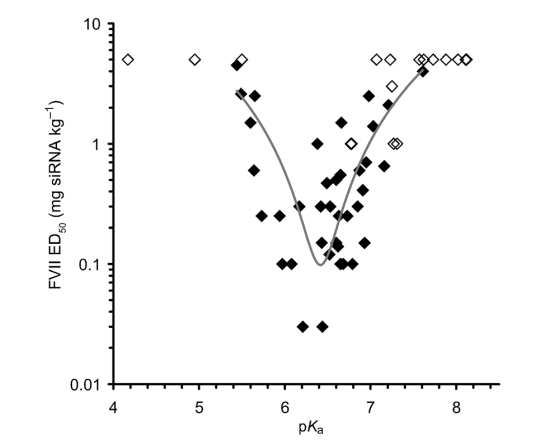
图2.2. 小鼠体内肝脏基因沉默活性与pKa的关系图
将56个可电离阳离子脂质配制成LNP,并进行ED50分析,根据它们的pKa,绘制得到图4。有15种脂质,没有达到ED50剂量。剩余的脂质,通过最佳拟合曲线突出显示了最具活性的化合物,其表现出在6.2和6.5之间的最佳pKa
表2.1. 56种可电离阳离子脂质的设计及其ED50,pKa值考察
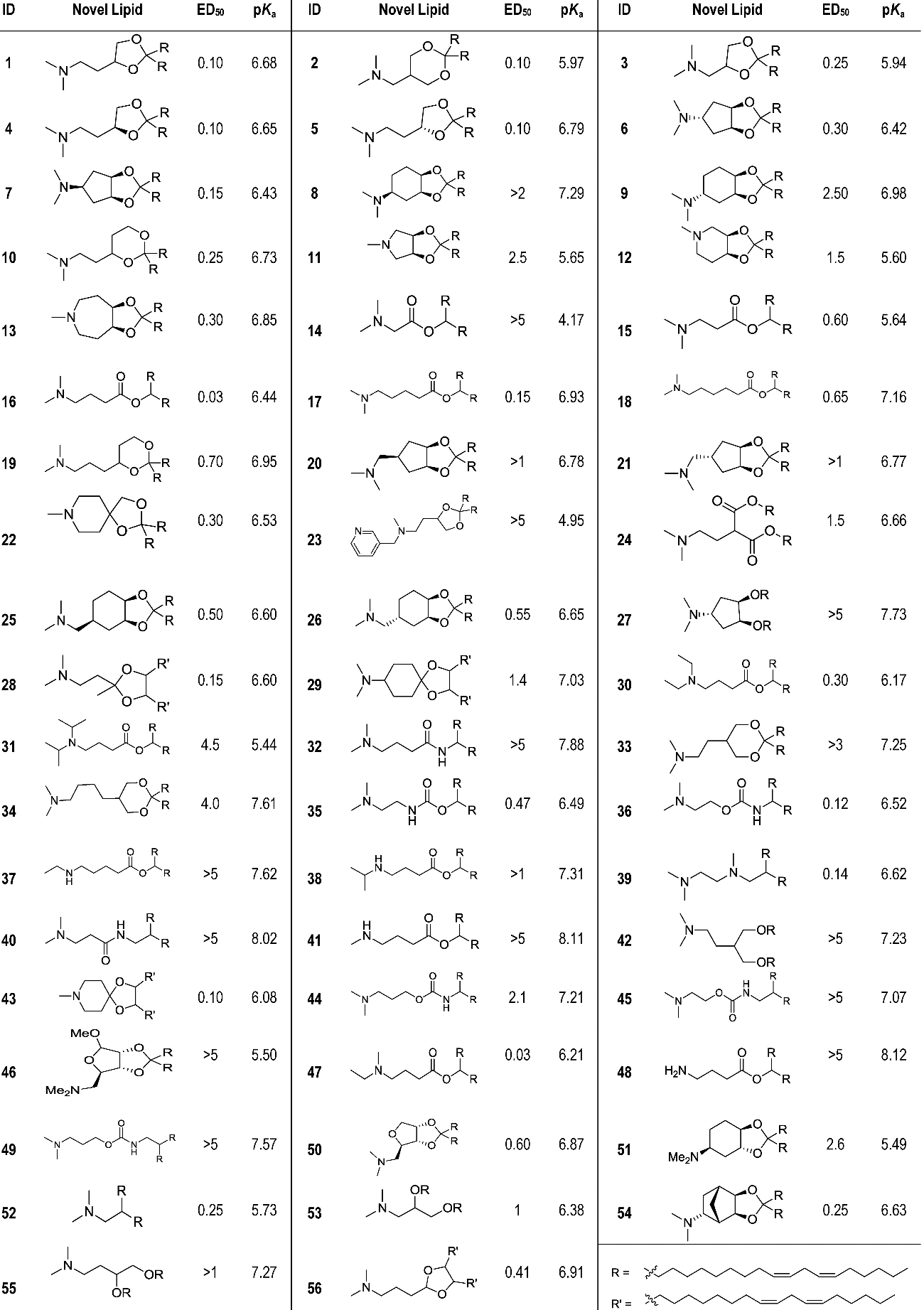
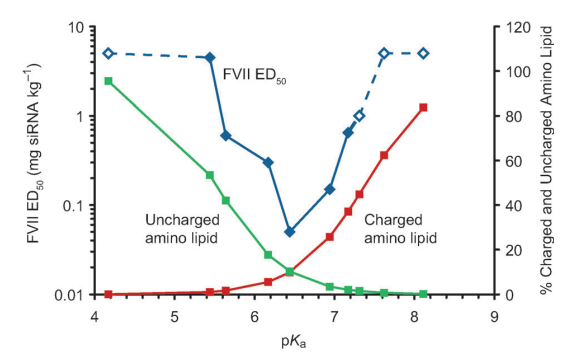
图2.3. 小鼠肝脏基因沉默活性图
pKa在4-8.5之间,计算质子化/未质子化可电离阳离子脂质的电离程度(%摩尔分数)。每个FVII ED50数据点(蓝色)来源于四剂量反应(每个剂量水平n=4只小鼠)。假设pH为7.4,在血液中携带正电荷的阳离子脂质显示为红色,而在pH为5.5的酸性内涵体中不带电的阳离子脂质的显示为绿色。
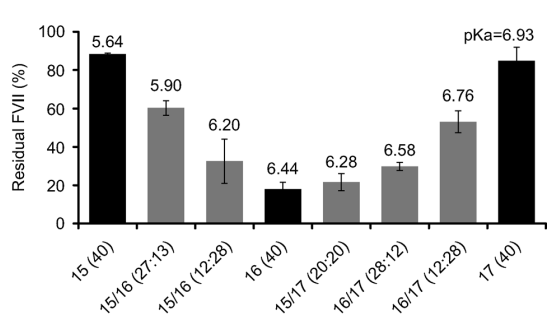
图2.4.支持pKa值作为决定可电离阳离子脂质,体内肝脏基因沉默活性的主导因素。
可电离阳离子脂质15、16和17的混合物使所得LNP的平均表面pKa值在5.64和6.93之间递增移位。括号中的数字表示混合物中阳离子脂质的摩尔比(氨基脂质组分)。
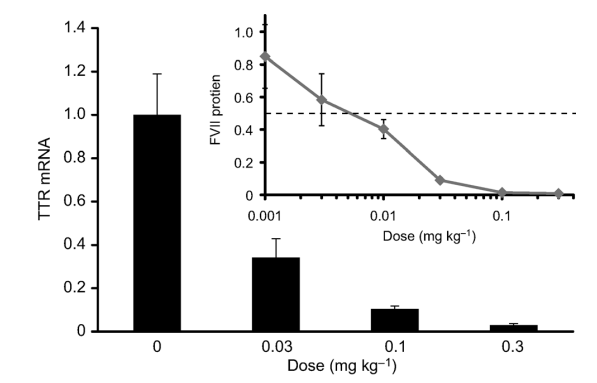
图2.5.可电离阳离子脂质(16,DLin-MC3-DMA)的siRNA-LNP在小鼠和非人类灵长类动物中的功效。
在本研究中使用了含有50摩尔%的16(DLin-MC3-DMA)的经过优化后的制剂组合。相对于具有约0.005 mg.kg-1的中值有效siRNA剂量(ED50)的生理盐水对照组,小鼠血清中的残余FVII活性;柱状图代表平均值(n=5)给予食蟹猴0.03、0.1和0.3mg.kg-1剂量的靶向TTR基因的siRNA。线型图则表示平均值(n=3),以TTR mRNA表示,相对于肝脏样本中测定的GAPDH mRNA水平,ED50估计为<0.03 mg.kg-1。

Reference
[1]. Semple SC, Akinc A, Chen J, Sandhu AP, Mui BL, Cho CK, Sah DW, Stebbing D, Crosley EJ, Yaworski E, Hafez IM, Dorkin JR, Qin J, Lam K, Rajeev KG, Wong KF, Jeffs LB, Nechev L, Eisenhardt ML, Jayaraman M, Kazem M, Maier MA, Srinivasulu M, Weinstein MJ, Chen Q, Alvarez R, Barros SA, De S, Klimuk SK, Borland T, Kosovrasti V, Cantley WL, Tam YK, Manoharan M, Ciufolini MA, Tracy MA, de Fougerolles A, MacLachlan I, Cullis PR, Madden TD, Hope MJ. Rational design of cationic lipids for siRNA delivery. Nat Biotechnol. 2010 Feb;28(2):172-6. doi: 10.1038/nbt.1602. Epub 2010 Jan 17. PMID: 20081866.
[2]. Jayaraman M, Ansell SM, Mui BL, Tam YK, Chen J, Du X, Butler D, Eltepu L, Matsuda S, Narayanannair JK, Rajeev KG, Hafez IM, Akinc A, Maier MA, Tracy MA, Cullis PR, Madden TD, Manoharan M, Hope MJ. Maximizing the potency of siRNA lipid nanoparticles for hepatic gene silencing in vivo. Angew Chem Int Ed Engl. 2012 Aug 20;51(34):8529-33. doi: 10.1002/anie.201203263. Epub 2012 Jul 10. PMID: 22782619; PMCID: PMC3470698.